P4Life: Signalwege
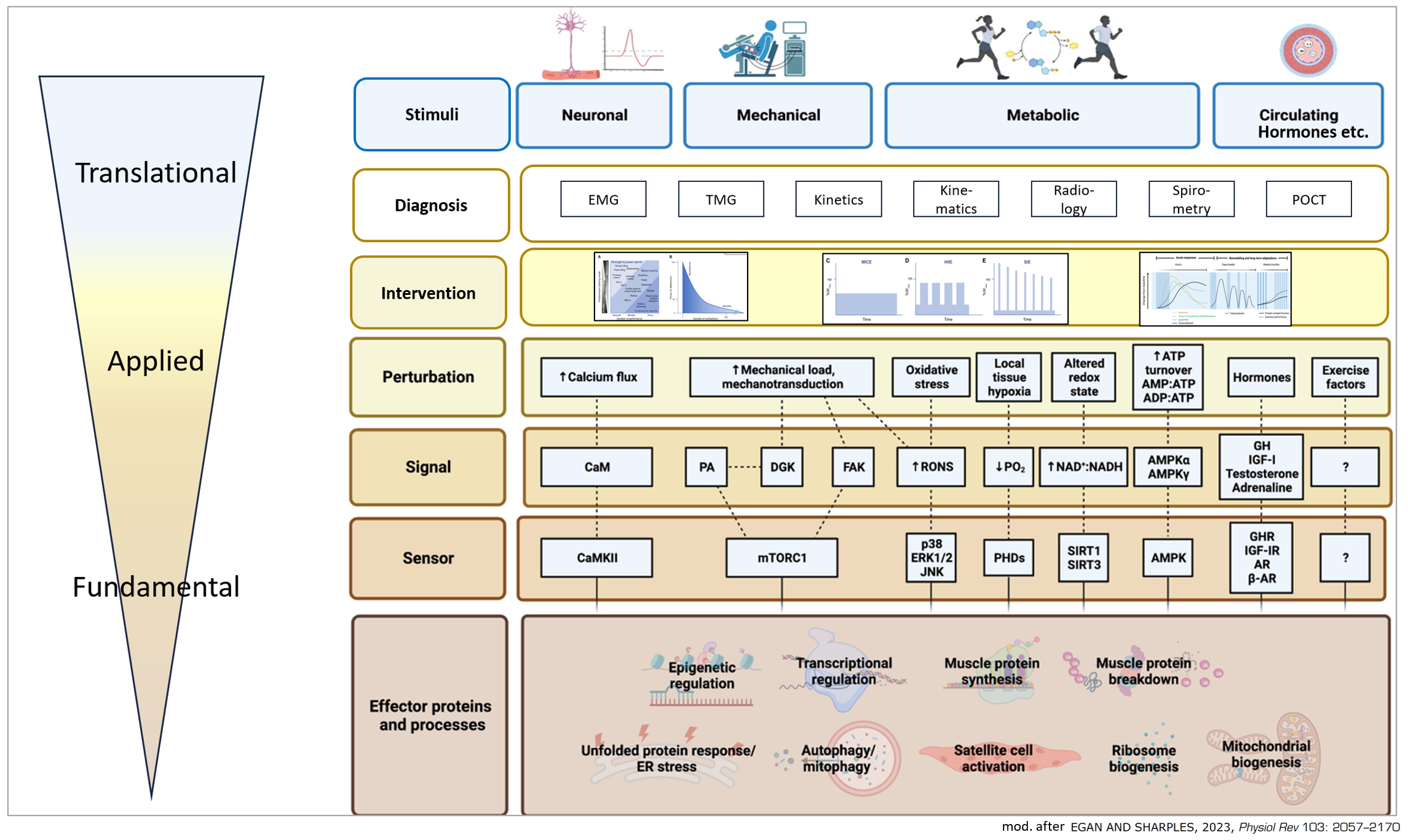
Die Abbildung veranschaulicht ein integratives Modell der molekularen Anpassungsprozesse in der Skelettmuskulatur an körperliche Belastung. Sie basiert auf der systematischen Gliederung von Stimuli über molekulare Signalwege bis hin zu Effektprozessen, aufgeteilt in drei Forschungsebenen: translational, applied und fundamental.
Die oberste Ebene zeigt verschiedene Reiztypen – neuronal, mechanisch, metabolisch sowie durch zirkulierende Hormone vermittelt. Darunter folgen diagnostische Methoden (z. B. EMG, Kinematik, Spiroergometrie), mögliche Trainingsinterventionen, gefolgt von den zellulären Perturbationen, die durch das Training ausgelöst werden (z. B. Kalziumflux, mechanische Last, oxidativer Stress, hypoxisches Gewebe, ATP-Umsatz etc.).
Diese führen zur Aktivierung spezifischer Signaltransduktionsmoleküle wie CaM, FAK, AMPK oder RONS, welche wiederum von Sensoren wie CaMKII oder mTORC1 erfasst werden. Daraus resultieren tiefgreifende zelluläre Anpassungsprozesse: Genexpression (z. B. durch epigenetische oder transkriptionale Regulation), Proteinsynthese, Ribosomen- und Mitochondrienbiogenese, Satellitenzellaktivierung sowie der Abbau nicht benötigter Strukturen.
Der Ansatz betont insbesondere: Die Bedeutung wiederkehrender, akuter molekularer Reaktionen auf Einzelbelastungen als Grundlage langfristiger Anpassung Die Beteiligung mehrerer Regulationsstufen: von Signalwegen über Transkriptions- und Translationsregulation bis zu Proteomumbau Die Rolle spezifischer Sensoren (z. B. AMPK, SIRT1, HIF-1α) für metabolische und hypoxische Zustände Die Notwendigkeit eines systembiologischen Verständnisses: Anpassung ist das Resultat der Integration multipler Signalwege, nicht einzelner „Masterregulatoren“ Diese Visualisierung zeigt somit nicht nur eine Momentaufnahme molekularer Prozesse, sondern betont deren Bedeutung für die Langzeitadaption des Muskels durch wiederholte Trainingsreize. Sie dient zugleich als Framework für personalisierte Target-Strategien.
Eine besondere Herausforderung für die Forschung besteht darin, dass auf der genetischen Ebene der Bildung von Effektorproteinen die Zuordnung zu den Signalwegen an Klarheit verliert.
Ausgewählte Literatur
- Balagué, Natàlia; Hristovski, Robert; Almarcha, Maricarmen; Garcia-Retortillo, Sergi; Ivanov, Plamen Ch (2020): Network Physiology of Exercise: Vision and Perspectives. Frontiers in Physiology, 11, 611550.
- Egan, Brendan; Sharples, Adam P. (2023): Molecular responses to acute exercise and their relevance for adaptations in skeletal muscle to exercise training. Physiological Reviews, 103(3), 2057–2170.
- Furrer, Regula; Hawley, John A.; Handschin, Christoph (2023): The molecular athlete: exercise physiology from mechanisms to medals. Physiological Reviews, 103(3), 1693–1787.
- Iolascon, Giovanni; Gimigliano, Francesca; Di Pietro, Gioconda; Moretti, Antimo; Paoletta, Marco; Rivezzi, Matteo et al. (2021): Personalized paths for physical activity: developing a person-centered quantitative function to determine a customized amount of exercise and enhancing individual commitment. BMC Sports Science, Medicine & Rehabilitation, 13(1), 60.
- López-Otín, Carlos; Kroemer, Guido (2021): Hallmarks of Health. Cell, 184(1), 33–63.
